在网站 http://mapofcpan.org/ 上有一个非常好看的对CPAN Perl模块可视化的例子,如下图:
在这篇文章中,我将展示如何使用R包HilbertCurve来绘制这样的图。
我们首先读入CPAN上所有模块的名字,这个可以在 https://www.cpan.org/modules/02packages.details.txt 中找到。下面我们直接将这个文件读入:
df = read.table("https://www.cpan.org/modules/02packages.details.txt", skip = 9)
head(df)
## V1 V2 V3
## 1 A1z::Html 0.04 C/CE/CEEJAY/A1z-Html-0.04.tar.gz
## 2 A1z::HTML5::Template 0.22 C/CE/CEEJAY/A1z-HTML5-Template-0.22.tar.gz
## 3 A_Third_Package undef C/CL/CLEMBURG/Test-Unit-0.13.tar.gz
## 4 AAA::Demo undef J/JW/JWACH/Apache-FastForward-1.1.tar.gz
## 5 AAA::eBay undef J/JW/JWACH/Apache-FastForward-1.1.tar.gz
## 6 AAAA undef P/PR/PRBRENAN/Data-Table-Text-20210818.tar.gz
模块的名字在第一列中,我们首先将其排序。
all_modules = sort(df[, 1])
我们取每个模块的“命名空间”。命名空间是第一个::前面的字符串。注意,在Perl中,这可能不叫做命名空间,在这篇文章,为方便起见,我们称之为命名空间。
ns = gsub("::.*$", "", all_modules)
现在ns是一个字符串向量,下面我们将其中的每一个值对应到一条Hilbert曲线上。我们首次将其转换为一个Rle对象。
library(IRanges)
r = Rle(ns)
Rle是一个很简单的类,定义在IRanges包中。在变量r中,每一个元素对应着ns中一个单独的命名空间,在向量ns中,每一个命名空间的位置和长度也被计算出。在下面的代码中,我们提取出这些信息:
s = start(r) # start position of each ns in r
e = end(r) # end position of each ns in r
w = width(r) # width of each ns in r
labels = runValue(r) # corresponding labels
现在我们可以进行Hilbert曲线可视化了。我们将最大的30个命名空间高亮和加上名字。
library(HilbertCurve)
ind = order(w, decreasing = TRUE)[1:30]
w_cutoff = w[ ind[30] ]
set.seed(123) # because we have randomm colors
hc = HilbertCurve(0, length(all_modules), level = 8, mode = "pixel")
hc_layer(hc, x1 = s, x2 = e,
col = circlize::rand_color(nrun(r), transparency = ifelse(w >= w_cutoff, 0, 0.8)))
hc_text(hc, x1 = s[ind], x2 = e[ind], labels = labels[ind],
gp = gpar(fontsize = 9))
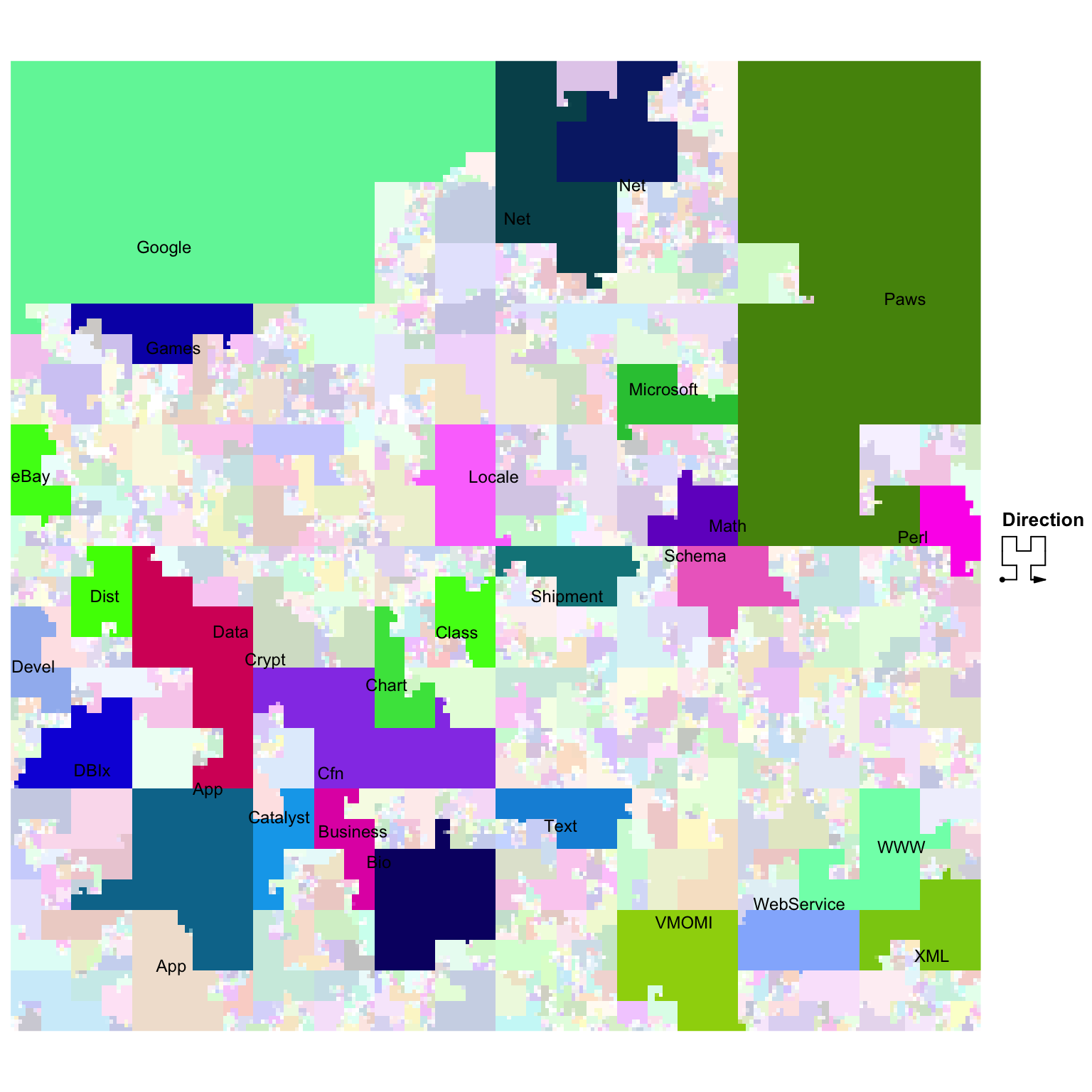
注意,部分文字的位置不是非常好,这以后可以改进。
可能会有人会说,这个其实就是可视化不同类别中软件包的数目,本质上和barplot一样。其实只说对了一半。的确,可视化的信息和barplot是一样的, 但是barplot本质上只是一维上的可视化,而Hilbert曲线是一种”二维“上的可视化技术。如果非要找一种类似的可视化技术,那么应该和Bubble chart比较类似 https://r-graph-gallery.com/circle-packing.html 。
sessionInfo()
## R version 4.4.2 (2024-10-31)
## Platform: aarch64-apple-darwin20
## Running under: macOS 26.1
##
## Matrix products: default
## BLAS: /Library/Frameworks/R.framework/Versions/4.4-arm64/Resources/lib/libRblas.0.dylib
## LAPACK: /Library/Frameworks/R.framework/Versions/4.4-arm64/Resources/lib/libRlapack.dylib; LAPACK version 3.12.0
##
## locale:
## [1] C.UTF-8/UTF-8/C.UTF-8/C/C.UTF-8/C.UTF-8
##
## time zone: Europe/Berlin
## tzcode source: internal
##
## attached base packages:
## [1] grid stats4 stats graphics grDevices utils datasets
## [8] methods base
##
## other attached packages:
## [1] HilbertCurve_2.5.1 IRanges_2.40.1 S4Vectors_0.44.0
## [4] BiocGenerics_0.52.0 knitr_1.50 colorout_1.3-2
##
## loaded via a namespace (and not attached):
## [1] httr_1.4.7 cli_3.6.4 rlang_1.1.5
## [4] xfun_0.51 png_0.1-8 circlize_0.4.16
## [7] UCSC.utils_1.2.0 jsonlite_1.9.0 colorspace_2.1-1
## [10] htmltools_0.5.8.1 GlobalOptions_0.1.4 sass_0.4.9
## [13] rmarkdown_2.29 evaluate_1.0.3 jquerylib_0.1.4
## [16] fastmap_1.2.0 GenomeInfoDb_1.42.3 yaml_2.3.10
## [19] lifecycle_1.0.4 bookdown_0.44 compiler_4.4.2
## [22] Rcpp_1.0.14 XVector_0.46.0 blogdown_1.19
## [25] digest_0.6.37 R6_2.6.1 shape_1.4.6.1
## [28] polylabelr_0.3.0 GenomeInfoDbData_1.2.13 GenomicRanges_1.58.0
## [31] bslib_0.9.0 tools_4.4.2 zlibbioc_1.52.0
## [34] cachem_1.1.0
本文使用 CC BY-NC-SA 4.0 协议发布。